

Changes in soil organisms in crop rotation farmland accessed by DNA metabarcoding
Toward elucidating the interactions among soil environment, plants, and soil organisms Toshihiko Eki
A research team led by Professor Toshihiko Eki from the Department of Applied Chemistry and Life Science, Toyohashi University of Technology (concurrently posted with the Research Center for Agrotechnology and Biotechnology) used DNA metabarcoding to analyze changes in the composition of soil organisms associated with crop growth in two corn-cabbage rotation fields in Tahara City. The results showed that the soil biota and their networks in agricultural land are unexpectedly easily altered by the soil environment, cultivation history and crops. In the future, the DNA metabarcoding for soil biota analysis will enable the elucidation of the relationship between crops and soil organisms, as well as the evaluation of the biological properties of farmland and prediction of crop diseases, which are expected to contribute to improved crop cultivation technologies.
Historically, humans have been connected to the soil through agriculture. In recent years, there has been a growing movement internationally to reaffirm the importance of soil and its conservation, as can be seen with the designation of the year 2015 as the International Year of Soil. Diverse organisms such as bacteria, nematodes, arthropods, and fungi exist in soil and form a soil ecosystem, but their actual compositions and dynamics are unclear. Previously, the research team analyzed nematodes by fully utilizing the DNA barcoding, which identifies biological species based on differences in the organism’s unique nucleotide sequences in genes, revealing the unique nematode communities that are adapted to various soil environments. The research team has also introduced “DNA metabarcoding” using a next-generation sequencer that can decode vast quantities of nucleotide sequences, to analyze all of the prokaryotes and eukaryotes living in soil.
In the present study, the research team applied the DNA metabarcoding to the soil of two fields in Tahara City where corn and cabbage were rotated in 2019 (Field 1 and Field 2) to investigate the taxonomic composition and diversity of pro- and eukaryotes associated with crop growth, their networks, and the correlation between soil chemical factors and organisms. The two fields targeted for research had the following differing cultivation history: in the previous year (2018), Field 1 was plowed with green manure and not cultivated, whereas Field 2 had the same crop rotation of corn and cabbage. Parts of the 16S and 18S ribosomal RNA genes were used for DNA barcodes to classify pro- and eukaryotes, and a total of 3,086 eukaryotic sequence variants (SVs) and 17,069 prokaryotic SVs were identified as SVs with unique nucleotide sequences. A detailed analysis of the SV data showed firstly, that there were biological communities with different compositions in the four soils with different fields and crops, indicating that soil organisms were affected by both fields and crops. Secondly, the research team investigated the correlation between soil chemical factors and organisms, finding biological groups and major SVs that were closely correlated with soil pH, moisture content, nitrate nitrogen, nutrient salt concentrations, and other factors. Finally, a network analysis of pro- and eukaryotic SVs showed for the first time that the network structure in the corn-cultivated soil of Field 1, which was not cultivated in the previous year, was clearly less complex than the other soils, but that the network became more complex with subsequent cabbage cultivation. This process largely involved not only fungi but also protists such as Cercozoa. This research showed that the soil biota and biological networks in agricultural land are influenced and easily changed by the soil environment (e.g., chemical factors), cultivation history, and crops.
This research showed that DNA metabarcoding is a useful approach for the biological analysis of agricultural soils. Recent research has shown that the gut microbiota influences human health, and the relationship between plants (crops) and soil organisms can be compared to the relationship between humans and their gut microbiota. The research team believes that soil organisms, particularly those living in the rhizosphere, are deeply involved in the health and growth of crops, and they would like to develop their research in the future by focusing on the interactions between plants and rhizosphere organisms.
This research was supported by the Takahashi Industrial and Economic Research Foundation and the JSPS Grant-in-Aid for Scientific Research (KAKENHI) (grant number: 22H02326). The research team would also like to extend their sincere gratitude to Project Associate Professor Takahiro Yamauchi of the Research Center for Agrotechnology and Biotechnology and Kihachiro Yamamoto of Tahara City for their considerable assistance in this research.
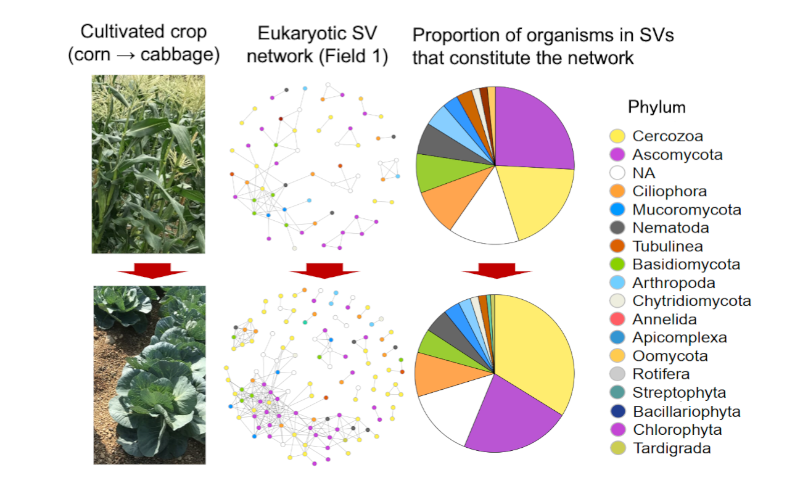
Changes in network of eukaryotic SVs (center) and proportion of SVs that constitute the network (right) due to the shift in cultivation from corn to cabbage (left, top to bottom)
Reference
Harutaro Kenmotsu, Tomoro Masuma, Junya Murakami, Yuu Hirose and Toshihiko Eki (2023). Distinct prokaryotic and eukaryotic communities and networks in two agricultural fields of central Japan with different histories of maize-cabbage rotation. Scientific Reports, 13(1): 15435.
doi:10.1038/s41598-023-42291-y
DNAを通して見た輪作農地における土壌生物の変動
土壌環境、植物、土壌生物間相互作用の解明に向けて 浴 俊彦豊橋技術科学大学応用化学・生命工学系(先端農業・バイオリサーチセンター兼務)浴 俊彦教授らの研究チームは、DNAメタバーコード法を用いて、田原市の2箇所のトウモロコシ・キャベツ輪作畑において、作物成長に伴う土壌生物の変動を解析しました。その結果、農地の土壌生物相や生物間ネットワークは、想像以上に、土壌環境、耕作履歴や作物による複合的な影響を受け、容易に変化することが明らかになりました。今後、土壌生物相解析法を活用することで、作物と土壌生物の関係が解明されるとともに、農地の高精度な生物性評価や作物病害予測が可能となり、作物栽培の技術改善に貢献することが期待されます。
土壌と人類とは、歴史的に農業を通じて、互いに深く関わってきました。2015年が国際土壌年に制定されたように、近年は国際的に土壌の重要性とその保全を再認識する動きが盛んになっています。土壌には、細菌や線虫、菌類など多様な生物が存在し、土壌生態系を構築していますが、その実態は明らかではありません。これまで研究チームは、遺伝子における生物固有の塩基配列の違いに基づいて生物種を識別する「DNAバーコード法」を駆使して、線虫の分析を行い、様々な土壌環境に適応した線虫集団の存在を明らかにしてきました。また、膨大な塩基配列を解読できる次世代シークエンサーを用いた「DNAメタバーコード法」を確立し、土壌に生息するすべての原核生物と真核生物の分析を可能にしました。
本研究では、2019年にトウモロコシとキャベツの輪作を行った、田原市の2箇所の畑(畑1と畑2)土壌に対して、DNAメタバーコード法を適用し、作物成長に伴う原核生物と真核生物の系統組成や多様性、生物間ネットワーク、土壌の化学性と生物の相関関係を調べました。研究対象の2つの畑には、前年の2018年に、畑1では、緑肥をすき込み、栽培を行わなかったのに対して、畑2では、同じトウモロコシ・キャベツの輪作を行ったという耕作履歴の違いがあります。原核生物と真核生物の分類のためのDNAバーコードには、16S, 18SリボソームRNA遺伝子の一部を使用し、塩基配列がわずかに異なる配列バリアント(sequence variant, SV)として、合計3,086種の真核生物SV、17,069種の原核生物SVを同定しました。SVのデータを詳しく解析した結果、第一に、畑と作物が異なる4つの土壌には組成の異なる生物群が存在し、畑や作物により土壌生物が影響を受けたことが示されました。第二に、土壌の化学性と土壌生物群との相関を調べ、土壌のpHや含水率、硝酸態窒素や栄養塩類の濃度等と密接に相関する生物系統やSVを見出しました。最後に、原核生物SV及び真核生物SVのネットワーク解析を行った結果、前年度に耕作を行わなかった畑1のトウモロコシ栽培時のネットワーク構造は、他と比べて、明らかに複雑度が低く、その後のキャベツ栽培により、ネットワークが複雑化したこと、この過程には菌類のほか、ケルコゾアなど原生生物が関与することを初めて明らかにしました。本研究により、農地の土壌生物相や生物間ネットワークは、想像した以上に、化学性などの土壌環境、耕作履歴や作物によって、複合的な影響を受け、容易に変化しうることが明らかになりました。
本研究から、DNAメタバーコード法が、農地の土壌生物分析に有用であることが示されました。近年の研究で、腸内細菌叢(そう)が人の健康に関与することが明らかにされつつありますが、植物(作物)と土壌生物との関係は、人と腸内細菌の関係に例えることができると考えられます。研究チームは、土壌生物、特に根圏の土壌生物が、作物の健康や生育に深く関与すると考え、今後、植物と根圏生物の相互作用に注目して、研究を展開したいと考えています。
本研究は、高橋産業経済研究財団、及びJSPS科研費(課題番号:22H02326)の支援を受けて実施しました。また、先端農業・バイオリサーチセンターの山内高広特任准教授と田原市の山本喜八朗氏には、本研究に多大なご協力をいただきました。この場をお借りして、感謝申し上げます。
Researcher Profile

| Name | Toshihiko Eki |
|---|---|
| Affiliation | Department of Applied chemistry and Life Science |
| Title | Professor |
| Fields of Research | Molecular Genetics / Molecular Biology / Functional Genomics |
