

3 Million Cells per Minute: Parallel Microdevice with AI-Powered Single-Cell Analysis
Moeto Nagai
Researchers at the Indian Institute of Technology (IIT) Madras and Toyohashi University of Technology (TUT) have developed an integrated platform that advances both fronts simultaneously. The platform combines two modules: a massively parallel through‑hole cell‑squeezing mechanoporation device for high‑throughput intracellular delivery, and an automated single‑cell image‑cytometry pipeline built on Mask R‑CNN.
Many applications—from drug discovery and diagnostics to cell engineering and gene modulation—require delivering biomolecules into large numbers of cells and rapidly evaluating the outcomes. The challenge is twofold: achieve intracellular delivery at scale across diverse cells and cargos, and obtain quantitative results fast enough to keep pace with that delivery.
The device guides cells through an array of up to 62,000 tiny through-holes that are narrower than the cell diameter. A brief, gentle squeeze creates transient membrane pores that admit biomolecules into the cell interior, then reseal, allowing cells to recover. In validation studies, the team delivered gene‑silencing RNA (siRNA) and plasmid DNA across multiple cell types, including human gingival fibroblasts (hGFs), demonstrating broad utility for cell engineering and personalized therapies.
To keep up with the fast delivery, an automated analysis system uses the same microscope images labs already acquire. A deep‑learning model looks at those images and, in a single pass, reports four readouts: cell size, the fraction of cargo‑positive cells, the fraction of viable cells, and per‑cell fluorescence intensity. What used to take hours of manual counting now takes about 83 seconds on a representative dataset of roughly 500 cells, with accuracy comparable to human review. Large cohorts were processed—for example, 1,980 cells (6‑FAM siRNA) and 1,184 (EGFP plasmid). These sample sizes make the results statistically robust and turn high‑throughput delivery into high‑confidence decisions.
“Our goal was simple: get molecules inside many cells quickly and gently,” says first author Pulasta Chakrabarty. “Seeing the device work across different cell types points to real potential in cell engineering and personalized therapies.” Co‑author Ryoma Suzuki adds, “The automated model looks at the same images labs already use. It does the counting and measuring—cell size, delivery efficiency, viability, and per‑cell fluorescence—in one pass, so the evaluation keeps pace with the experiments.” “High throughput alone isn't enough,” notes Prof. Moeto Nagai. “What matters is the speed at which results can be trusted. By unifying high‑throughput delivery with automated quality control, we move from proofs‑of‑concept to practical workflows—and closer to systems that prepare a patient’s cells on‑site.”
By coupling scalable intracellular delivery with rapid, automated evaluation, the platform expands what can be accomplished in a single day—from large‑scale screening and diagnostics to practical cell manipulation and future point‑of‑care gene‑editing workflows.
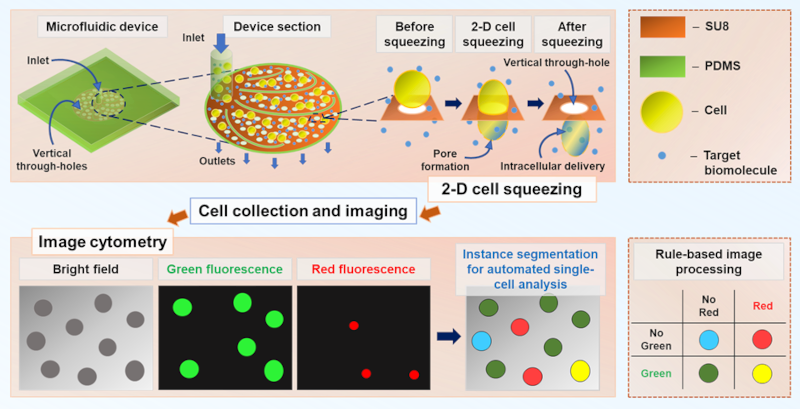
Top row (delivery and imaging). Cells and cargo are introduced into a microfluidic chip whose base integrates a porous SU‑8 membrane with vertical through‑holes and a PDMS chamber. As individual cells pass the sub‑cell‑sized through-holes, a brief 2D squeeze forms transient membrane pores, allowing cargo to enter; after passage, the membrane reseals, and cells exit to the outlets. Treated cells are then collected and imaged under a microscope (bright‑field and fluorescence).
Bottom row (automated single‑cell analysis). The same image set is processed by image cytometry using a Mask R‑CNN–based instance‑segmentation model: (i) bright‑field for cell localization, (ii) green fluorescence for cargo delivery, (iii) red fluorescence for viability/dying cells (e.g., PI or Calcein red‑orange), and (iv) instance‑segmentation overlays for per‑cell measurements. A simple rule-based map classifies each cell by the presence/absence of green and red signals into four states (delivered‑live, delivered‑dead, undelivered‑live, undelivered‑dead), enabling automatic calculation of delivery efficiency, viability, and per‑cell delivered amount. The diameter of each cell in an image is evaluated from the instance segmentation masks.
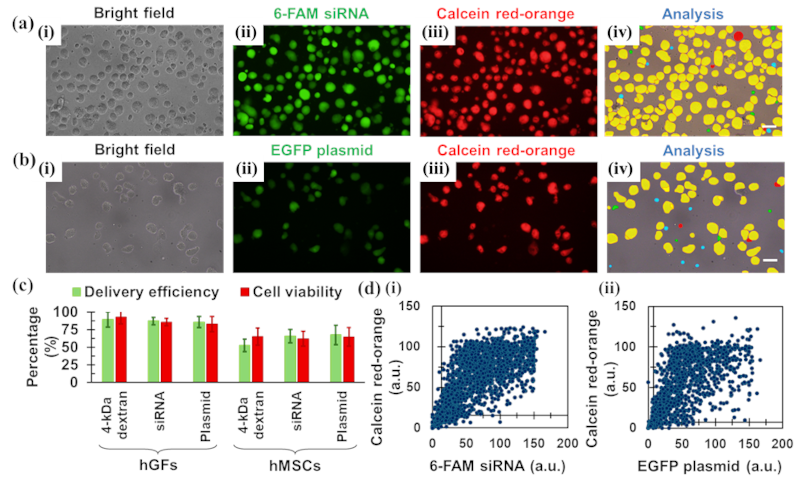
(a) 6‑FAM siRNA; (b) EGFP plasmid. For each condition, (i) bright‑field to locate cells, (ii) green fluorescence reporting intracellular cargo, (iii) Calcein red‑orange as the live/dead readout, and (iv) image‑cytometry overlays showing per‑cell segmentation and delivery/viability class. Scale bar: 20 µm.
(c) Summary bars of delivery efficiency (green‑positive fraction) and cell viability (Calcein‑positive fraction), reported as mean ± SD across fields/replicates.
(d) Single‑cell scatter plots linking signal to state: (i) 6‑FAM siRNA (N=1,980) and (ii) EGFP plasmid (N=1,184). X‑axis, green fluorescence (a.u., delivered amount proxy); Y‑axis, Calcein red‑orange (a.u., viability proxy). These panels connect raw images to cohort metrics and per‑cell quantification in one workflow.
Reference
“High Throughput Intracellular Delivery Using a 2D Cell-Squeezing Mechanoporation Device and Its Analysis by a Deep Learning Model”
Pulasta Chakrabarty, Abinaya R, Ryoma Suzuki, Srikanth Vedantam, Suresh Rao, Moeto Nagai, Tuhin Subhra Santra
First published: 21 August 2025
https://doi.org/10.1002/adhm.202502472
6万個微小穴の並列デバイスで毎分300万個の細胞に分子送達、AI画像解析で単一細胞評価を同時実現
永井萌土豊橋技術科学大学とインド工科大学マドラス校の共同研究チームは、直径8–15 µm(8 µm時に最大約62,000孔)、深さ約20 µmの垂直スルーホールを持つマイクロ流体デバイスを開発しました。最大毎分300万個の細胞処理能力(細胞濃度2×10⁶ cells/mL、流量1.5 mL/min条件下)を実現し、深層学習(Mask R‑CNN)による自動画像解析と組み合わせることで、細胞内送達と評価を同時に高速化するプラットフォームを実現しています。多様な細胞に分子を素早く細胞内まで届け、その結果を数千単位の細胞で即座に数値化できます。
創薬や診断、細胞工学の現場では、膨大な数の細胞に対して分子を届け、その反応を同時に評価する「ハイスループット」な実験が不可欠です。ところが、従来のチップは処理速度や対応できる細胞・分子の幅に限界があり、結果の判定にも高価で複雑な装置や人の目が必要でした。本研究は、「速く届ける」と「速く確かめる」をひとつの流れにまとめ、ボトルネックを同時に解消しました。
デバイス底面の多孔膜(SU 8)には、細胞より細い垂直スルーホールが多数並びます。細胞がここを通過する一瞬だけ、穏やかに圧縮され、膜に一過性のナノ孔が開き、分子が細胞内へ入ります。孔は自然に閉じ、細胞は回復します。実験後は、顕微鏡画像(明視野・蛍光)を撮影して、オフラインで深層学習(Mask R‑CNN)ベースのイメージサイトメトリが解析し、送達効率、生存率、細胞サイズ、細胞あたりの送達量までを自動算出します。手作業なら数時間かかる処理が、約500細胞の代表的データセットでは約83秒で完了します。人力での評価と同等の精度を維持しています。
本プラットフォームは、HeLa細胞やJurkat細胞への4–40 kDaのFITC-デキストラン導入に加え、ヒト歯肉線維芽細胞(hGFs)やヒト間葉系幹細胞(hMSCs)など複数の細胞で、蛍光標識siRNA(6 FAM siRNA)やEGFPプラスミドDNAの細胞内送達を確認しました。1,980細胞(siRNA)や1,184細胞(プラスミド)といったサンプル数での解析により、統計的に信頼できる結果を短時間で得られます。AIの判定精度は人での評価と同程度で、解析時間は大幅に短縮されました。
研究者の声
「我々の目標はシンプルでした。たくさんの細胞の中へ、分子を“速く、穏やかに”届けることです。複数の細胞種で確かめ、個別化医療や細胞工学への可能性が広がりました」(筆頭著者 インド工科大学マドラス校 博士課程 Pulasta Chakrabarty)
「研究現場で普段撮っている画像を、そのままAIに渡すだけで解析できます。輝度に基づいて、送達効率、生存率、細胞サイズ、細胞あたりの量まで一度に数えます。細胞実験が進むスピードに、評価の方が追いつきます」(共著者 豊橋技術科学大学 機械工学専攻博士前期課程 鈴木涼真)
「ハイスループットでの物質送達と自動での品質管理を一体化して、概念実証から実務的なワークフローに橋渡しを行いました。将来的には患者さんの細胞を現場で処理し、条件探索を迅速化できるシステムへの応用を目指しています」(豊橋技術科学大学 次世代半導体・センサ科学研究所 永井萌土 教授)
意義と展望
1回通過の高スループットでの細胞処理を短時間に連続実行し、同一の撮像・AI解析パイプラインで多条件・多細胞種・多様な分子を横断比較が可能です。実験設計の自由度が広がります。
解析の自動化により、撮像直後に指標を可視化でき、その日のうちに条件決定が可能です。スクリーニングや診断、実用的な細胞改変、将来的なポイントオブケア型の遺伝子編集キットにつながる可能性があります。
今後は、より臨床に近い初代培養細胞や、タンパク質・ナノ粒子など多様な分子への適用拡張、並びに 装置とAIの普及を目指します。
Researcher Profile

| Name | Moeto Nagai |
|---|---|
| Affiliation | Department of Mechanical Engineering |
| Title | Professor |
| Fields of Research | BioMEMS / Biohybrid System / Micro-Nano Mechatronics / MicroTAS / Biofabrication / Micromachining |
