
ここからコンテンツです。

DNA barcodes decode the world of soil nematodes
To understand soil ecosystems and contribute to advanced agriculture Toshihiko Eki
Professor Toshihiko Eki, a member both of Toyohashi University of Technology’s Department of Applied Chemistry and Life Sciences, and the Research Center for Agrotechnology and Biotechnology, led a research team which used a next-generation sequencer to develop a highly efficient method to analyze soil nematodes. This method focused on using the 18S ribosomal RNA gene regions as DNA barcodes. They successfully used this method to reveal characteristics of nematode communities that inhabit fields, copses, and home gardens. In the future, the target will be expanded to cover all soil-dwelling organisms in agricultural soils, etc., to allow accurate investigations into a soil’s environment and bio-diversity. This is expected to contribute to the advancement of agriculture.
The United Nations (UN) declaration of 2015 as the ‘International Year of Soil’, was symptomatic of a worldwide push to raise awareness of the vital role played by the soil that covers our Earth, and the need to preserve it. Diverse groups of organisms such as bacteria, fungi, protists, and small soil animals inhabit the soil, and together they form the soil ecosystem. Nematodes are a representative soil animal; they are a few millimeters long and have a shape resembling a worm. They play an important role in the cycling of soil materials.
Many soil nematodes are bacteria feeders, but they have a wide variety of feeding habits, such as feeding on fungi, plant parasitism, or being omnivorous. In particular, plant parasitic nematodes often cause devastating damage to crops. Therefore, the classification and identification of nematodes is also important from an agricultural standpoint. However, nematodes are diverse, and there are over 30,000 species. Additionally, because nematodes resemble one another, morphological identification of nematodes is difficult for anyone but experts.
The research team focused on "DNA barcoding" to identify the species based on their unique nucleotide sequences of a barcode gene, and they established a method using a next-generation sequencer that can decode huge numbers of nucleotide sequences. They used this method to analyze nematode communities from different soil environments. Initially, four DNA barcode regions were set for the 18S ribosomal RNA genes shared by eukaryotes.
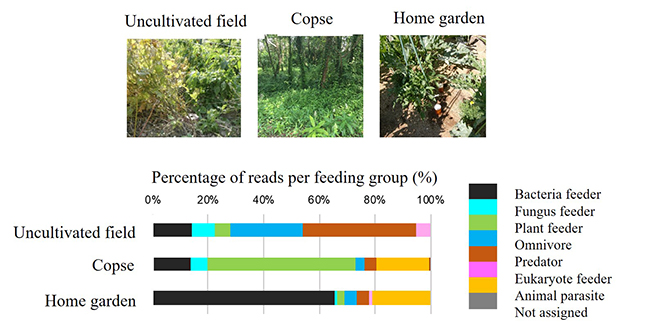
The soil nematodes used for analysis were isolated from an uncultivated field, a copse and a zucchini-cultivated home garden. The PCR was used to amplify the four gene fragments from the DNA of the nematodes and determine the nucleotide sequences. Additionally, the nematode-derived sequence variants (SVs) representing independent nematode species were identified, and after taxonomical classification and analysis of the SVs, it was revealed that plant parasitizing nematodes were more prevalent in the copse soil whereas bacteria feeders were more prevalent in the garden soil. It was also determined that predatory nematodes and omnivorous nematodes were abundant in the uncultivated field, in addition to bacteria feeders.
This DNA barcoding method using a next-generation sequencer is widely used for the analysis of intestinal microbiota, etc., but analyses of eukaryotes such as nematodes are still in the research stage. This research provides an example of its usefulness for the taxonomic profiling of soil nematodes.
Research team leader Professor Toshihiko Eki stated,
"In my work on genetic research, I have been dealing with nematodes (mainly C. elegans) for around 20 years. I came up with this theme as a suitable candidate for agriculture based research on behalf of our university's Research Center for Agrotechnology and Biotechnology. As a test, we isolated nematodes from the university's soybean field and an unmanaged flowerbed and analyzed the DNA barcode for each nematode. I found that in contrast to the soybean fields, which were dominated by bacterivorous nematodes, the flower beds were dominated by plant-parasitic nematodes that parasitize weeds and nematodes that prey on them.
This discovery was the start of our research (Morise et al., PLoS ONE, 2012). If we consider this method of using one-by-one DNA sequencing as being the first generation, the current method of using the next-generation sequencer can be considered as the second generation. With this method we were successfully able to clarify the characteristics of the nematode communities from each of the three soil environments."
Rather than isolating and analyzing soil nematodes, the research team is now developing a third-generation DNA barcoding method that directly extracts DNA from the soil and analyzes the entire organism inhabiting the soil. We are currently analyzing the soil biota of cabbage fields, and other areas. Our goal is to analyze how soil biota, including microorganisms, change with the growth of crops with a high degree of accuracy, to elucidate the relationship between crops and soil biota, and to clarify the effects of cultivated plants on soil biota and the biota associated with plant diseases at the genetic level.
If this research progresses, it will enable rational crop cultivation and management based on the results of soil organism analysis, which will contribute to the promotion of smart agriculture in Japan and overseas, including the Higashimikawa region, one of the most important agricultural areas in Japan.
This research was performed with the support of the Takahashi Industrial and Economic Research Foundation.
Reference
Harutaro Kenmotsu, Masahiro Ishikawa, Tomokazu Nitta, Yuu Hirose and Toshihiko Eki. Distinct community structures of soil nematodes from three ecologically different sites revealed by high-throughput amplicon sequencing of four 18S ribosomal RNA gene regions.
PLoS ONE 16(4), e0249571 (2021)
https://doi.org/10.1371/journal.pone.0249571
DNAバーコードが読み解く土壌線虫の世界
土壌生態系の解明やスマート農業の発展に向けて浴 俊彦
豊橋技術科学大学応用化学・生命工学系(先端農業・バイオリサーチセンター兼務)浴 俊彦 教授の研究チームは、次世代シークエンサーを活用し、18SリボソームRNA遺伝子領域をDNAバーコードとして用いた高精度な土壌線虫の分析法を開発しました。この方法を用いて、畑、雑木林、家庭菜園に生息する線虫集団の特徴を明らかにすることに成功しました。今後、対象を農地等の土壌生物全体に拡張することで、土壌環境や土壌生物多様性の高精度な調査が可能となり、農業のスマート化にも貢献できると考えられます。
国連が2015年を国際土壌年として設定したように、近年、地球を覆う土壌の重要性とその保全を再認識する動きが国際的に盛んになっています。土壌には、細菌、糸状菌、原生生物、土壌小動物など多種多様な生物群が生息し、それらが土壌生態系を形成しています。線虫は、体長数ミリのミミズに似た体型を持つ代表的な土壌動物であり、土壌物質の循環に重要な役割を果たしています。
多くの土壌線虫は、細菌を食べる細菌食性ですが、糸状菌食性、植物寄生性、雑食性など多様な食性を持つ線虫が存在し、とくに植物寄生性線虫は、作物の連作障害の原因にもなっています。そのため、農業の観点からも線虫の分類・識別は重要な課題ですが、線虫には3万種を超える種の多様性がある上、互いに形が似ているために線虫の形態による識別は、専門家でないと難しいのが実情です。
そこで、研究チームは、遺伝子における生物種固有の微小な塩基配列の違いに基づいて生物種を識別する「DNAバーコード法」に着目し、飛躍的な塩基配列解読能をもつ次世代シークエンサーを導入した手法を確立し、異なる環境土壌に生息する線虫集団の分析に応用しました。はじめに、真核生物が共通に持つ18SリボソームRNA遺伝子に4つのDNAバーコード領域を設定しました。
分析対象には、非耕作畑、雑木林、ズッキーニ栽培中の家庭菜園から分離した土壌線虫集団を用い、各線虫集団のDNAから、PCRで4つの遺伝子断片を増幅し、それらの塩基配列を解読しました。さらに、線虫に由来する塩基配列を抽出、系統ごとに分類・分析した結果、雑木林土壌では、植物寄生性線虫が、一方、家庭菜園土壌では、細菌食性線虫が多く生息していることを明らかにしました。また、非耕作畑には、細菌食性線虫のほか、動物捕食性や雑食性の線虫が多く存在することが判りました。
このような次世代シークエンサーを使ったDNAバーコード法は、原核生物である腸内細菌叢の分析などに広く応用されていますが、線虫などの真核生物を対象とした分析は、いまだ研究段階にあり、普及するには至っていません。今回の研究では、土壌線虫を例に、その有用性を示すことができました。
研究チームのリーダーである浴 俊彦教授は
「これまで、遺伝子研究を通じて、20年近く線虫と関わってきました。本学の先端農業・バイオリサーチセンターのメンバーとして、農業に関わる研究を何かできないかと思案する中で、このテーマを思いつきました。試みに、大学の大豆畑と(放置状態の)花壇から線虫を分離し、1頭ごとにDNAバーコード解析を行ったところ、細菌食性線虫が多かった大豆畑とは対照的に、花壇では、雑草に寄生する植物寄生性線虫と、それらを捕食する線虫で占められていることを発見したことがこの研究のスタートになりました(Morise et al., PLoS ONE, 2012)。この方法を第1世代とすると、今回は次世代シークエンサーを導入した第2世代の方法になりますが、期待通り、3つの土壌環境を反映した線虫集団の姿を明らかにすることができました。」 と述べています。
現在、研究チームは、土壌線虫を分離・分析するだけではなく、土壌から直接DNAを取りだして、そこに生息する生物全体をまとめて分析する、第3世代のDNAバーコード法を開発し、キャベツ畑などの土壌生物相の分析を進めています。作物の生長に伴い、微生物も含めた土壌生物集団がどのように変動するか高精度に分析し、作物と土壌生物群との関係を解明するとともに、栽培植物による土壌生物への影響や植物病害と関連する生物群を遺伝子レベルで明らかにすることを目指しています。
この研究が進めば、土壌生物の分析結果に基づいた合理的な作物栽培・管理が可能となり、日本でも有数の農業地帯である東三河地域を含めた国内外のスマート農業の推進に貢献できると考えています。
本研究は、公益財団法人 高橋産業経済研究財団のご支援を受けて実施されました。
Researcher Profile

| Name | Toshihiko Eki |
|---|---|
| Affiliation | Department of Applied Chemistry and Life Science |
| Title | Professor |
| Fields of Research | Molecular Genetics / Molecular Biology / Functional Genomics |
ここでコンテンツ終わりです。
